解説3:遺伝子破壊
担当:安達 佳樹
§概要
C. elegans の遺伝子破壊法としては、ターゲット遺伝子にトランスポゾン Tc1 が挿入した変異体を得る方法が実用化されている。Tc1 はミューテーター株において高頻度に転移し、各個体ごとに様々な部位へ挿入する。この株の集団中には、多くの遺伝子について Tc1 挿入変異体の存在が期待される。そこで、目的とするターゲット遺伝子の Tc1 挿入変異体を、集団中より選び出すことを行なう。
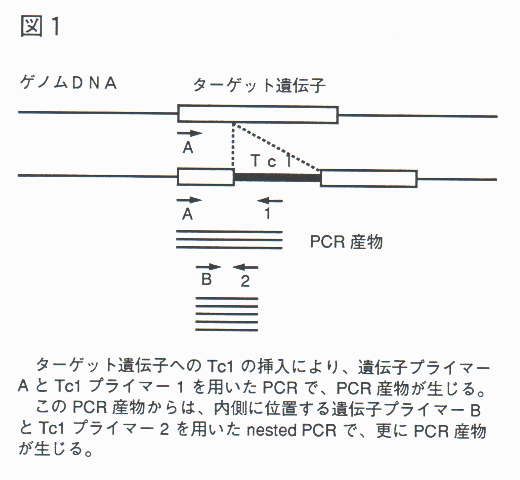
ターゲット遺伝子への Tc1 挿入変異は、ターゲット遺伝子と Tc1 のそれぞれの配列より作製したプライマーを用いた PCR により見い出すことができる(図1)。

すなわち、プライマーの位置関係が適当であれば、Tc1 挿入変異を持つ遺伝子からは PCR 産物が増幅され、これを目安とするわけである。
集団中より Tc1 挿入変異体を選び出すには、sib-selection 法を行なう。まず、少数個体より出発する培養を1世代以上行なう。培養集団の一部の個体よりゲノム DNA を抽出し、PCR により調べる。Tc1 挿入変異が確認されるなら、同じ Tc1 挿入変異を持つ同腹の個体(sibling)が培養集団の残りに存在することが考えられる。そこで、この残りをいくつかの小集団に分け、それぞれを培養する。これらを PCR により調べ、Tc1 挿入変異が存在する小集団については、更に同様のことを繰り返す。最終的に、Tc1 挿入変異を持つ1個体由来の集団を得る。
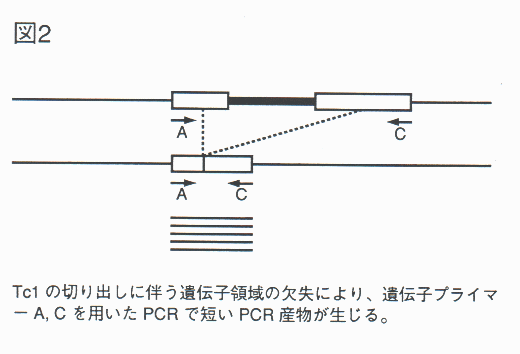
しかし、Tc1 挿入がターゲット遺伝子のイントロン内である場合、遺伝子機能を喪失していないことが考えられる。またエキソンへの挿入においても、遺伝子転写産物からスプライシングにより Tc1 配列が除かれ、mRNA からは機能を持つ蛋白産物が翻訳される場合があることも報告されている。(もちろんエキソンへの挿入により、遺伝子を破壊していることもある。)Tc1 は転移のためゲノム DNA から切り出される際、まれに挿入部位の近傍領域を欠失することが知られている。その結果エキソン領域を欠失すれば、確実に遺伝子を破壊していると期待される。そこで Tc1 挿入変異により遺伝子が破壊されていないと考えられる場合、Tc1 挿入変異体の集団中より更に欠失変異体を選び出すことを行なう。方法は Tc1 挿入変異体を得るものとほぼ同様であり、違いはミューテーター株自身の代わりに Tc1 挿入変異をホモ接合で持つミューテーター株を用いること、PCR で用いるプライマーは Tc1 挿入部位を挟む2ヶ所の遺伝子配列より作製し、PCR では野生株での増幅産物より短い増幅産物を目安とすることなどである(図2)。
§Tc1 挿入変異体の分離
Sib-selection 法で変異体を分離するには、培養集団中に同一の Tc1 挿入変異を持つ同腹の複数個体が存在しなければならない。ところが、Tc1 挿入変異は毎世代起こるため、培養最終世代の1個体に起こった Tc1 挿入変異も存在する。この場合、この個体のものを含むゲノム DNA より PCR 産物を得ることができても、同一の Tc1 挿入変異を持つ個体は集団の残りの内に存在しないため、この Tc1 挿入変異体を得ることはできない。そこで、集団中に同一の Tc1 挿入変異を持つ複数の個体が存在することを知るため、培養集団の複数の小集団からそれぞれゲノム DNA を抽出して PCR を行なう。これら全てにおいて同じ長さの産物が得られるならば、同一の Tc1 挿入変異を持つ個体が各小集団に存在することを示しており、培養集団の残りにもその Tc1 挿入変異を持つ個体が存在すると考えられる。
Tc1 挿入変異を見いだすための PCR において、一方のプライマーは Tc1 配列由来であり、Tc1 はゲノム当り数百コピー存在するので、非特異的な PCR 産物が生じ易い。そこで特異性を高めるため、この PCR 反応物を基に、更に内側プライマーを用いた PCR(nested PCR)を行なう(図1)。
ターゲット遺伝子領域への Tc1 挿入変異体が培養集団中に複数存在すると、遺伝子プライマーに最も近い Tc1 挿入変異由来の PCR 産物のみが見い出され、それ以外は隠されてしまうことがある。ここで問題となるのは、最終世代で起こった Tc1 挿入変異である。最終世代の個体数は多いので、この世代で生じる Tc1 挿入変異体も多い。ただ、このような Tc1 挿入変異は1個体のみに存在するので、複数個体に存在する Tc1 挿入変異に比べ、変異を持つゲノム DNA の絶対数は少ない。そこで、PCR に用いる DNA 量を少なくして、複数個体に存在した変異のゲノム DNA は数ゲノム分存在するが、1個体のみに存在した変異のゲノム DNA は確率的にほとんど無いような条件で PCR を行なう。
・Sib-selection
Sib-selection を行なう1例を示す。この方法の特徴は、多くの個体より出発する培養を1世代のみ行ない、培養開始時の個体に既に存在していた Tc1 挿入変異を選び出そうとするものである。
1)ミューテーター株より、成長ステージを揃えた虫集団を準備し、6cm シャーレに作製した NGM agarose plate - OP50 上に、約 600 個体を置く。
2)20度で餌がなくなるまで培養する。
3)培養集団を S basal 溶液中に集め、3等分する。
4)このうち2つ分の虫集団より S basal 溶液を除き、proteinase K 溶液で溶解する。(S basal 溶液が十分除かれていれば、 20ul 程度の proteinase K 溶液で溶解することができる。)
5)溶解液の 400 分の 1 量を用いて PCR を行なう。
6)PCR 反応液の 2,000 分の 1 量を用いて nested PCR を行ない、アガロースゲル電気泳動により分析する。
7)両虫集団から同じ長さの PCR 産物が得られた場合、残りの虫集団を約 25 匹ずつに分け、それぞれを培養する。
8)餌がなくなった時点で、各培養の虫の一部を proteinase K 溶液で溶解し、PCR(nested PCR は必要無いことが多い)で調べる。
9)PCR 産物が得られた場合、残りの虫より 1 匹から出発する培養を行ない、同様に調べる。
Tc1 挿入変異体を得るためのスクリーニングに必要な個体数は、遺伝子の大きさの違いなどにもよるが、30,000 匹程度が基準となる。これは、上記の方法で 50 プレート分に相当する。
PCR で見いだすことができる Tc1 挿入変異は、遺伝子プライマーより 2 kb 程度までに挿入したものである。ターゲット遺伝子が大きい場合、いくつかの領域に分けて別々に調べる。
Tc1 は2通りの方向で挿入するので、各向きの Tc1 プライマーを用いて別々に PCR を行なう。
PCR スクリーニングにおいて、複数(もしくは全て)の培養集団より同じ長さの PCR 産物が得られることがある。このような産物は、Tc1 挿入変異体の分離に結び付かないようである。
・ミューテーター株
MT3126 (mut-2 (r459) I; dpy-19 (n1347) III)
・NGM agarose plate - OP50
NGM agar の組成で、agar を agarose に変えたものである。agarose を用いる理由は、抽出するゲノム DNA に混入する酵素反応阻害物質が少ないことである。
OP50 はプレート全面に広げておく。
・proteinase K 溶液による虫体の溶解
組成 10 mM Tris・HCl (pH 8.0)
50 mM KCl
2.5 mM MgCl2
0.45 % Tween-20
0.05 % gelatin
500 ug / ml proteinase K
まず虫を凍結融解する。proteinase K 溶液を加え、(蒸発を防ぐためミネラルオイル溶液をのせ、)60度で1時間以上保温し、虫を溶解する。90度で15分間保温し、proteinase K を失活させる。
・PCR
組成 10 mM Tris・HCl (pH 8.3)
50 mM KCl
1.5 mM MgCl2
0.005 % gelatin
200 uM dATP, dGTP, dCTP, dTTP
0.4 uM プライマー
0.5 unit Taq polymerase
反応液量 20 ul
Tc1 プライマー(Tc1 全長 1,610 bp 配列 DDBJ - X01005)
Sequence Position Orientation
1 AGCCAGCTACAATGGCTTTC 336 - 317 Reverse
2 CCAAACAAATCCAGTGCAAC 251 - 232 Reverse
3 GATGCAAACGGATACGCGAC 1315 - 1334 Normal
4 TGTCATTTCCTTGCAACCTC 1396 - 1415 Normal
1,3が最初の PCR 用であり、2,4は nested PCR 用である。
反応条件
1)PCR 機器(PERKIN-ELMER / CETUS GeneAmp PCR System 9600 など)のヒートブロックを 94 度に熱しておき、反応液の入ったチューブを乗せる。
2)そのまま、1 分間保持する。
3)94 度 0.2 分、55 度 0.2 分、72 度 2.0 分のサイクルで行なう。
4)通常の PCR では 35 サイクル行ない、PCR + nested PCR では各 30 サイクル行なう。
§遺伝子欠失変異体の分離
Sib-selection 法を用いるので、複数個体に存在する欠失変異を見つけ出すことが必要である。そこで、培養集団中の複数の小集団由来の DNA からそれぞれ PCR を行ない、同じ長さの PCR 産物が得られるかを調べる。
PCR の2種類のプライマーは遺伝子由来であるので、PCR の特異性は高く、nested PCR は必要無い。
Tc1 の転移において、近傍領域の欠失を伴わない切り出しは比較的高頻度に起こる。それゆえ、PCR 産物として野生型遺伝子からの PCR 産物と同じ長さのものが必ず生じる。それで、この PCR 産物より短いものを目安とする。
・Sib-selection
Tc1 挿入変異体の分離法とほぼ同様である。
1)ターゲット遺伝子への Tc1 挿入変異をホモ接合に持つミューテーター株より、成長ステージを揃えた虫集団を準備し、 6cm シャーレに作製した NGM agarose plate - OP50 上に、約 600 個体を置く。
2)20度で餌がなくなるまで培養する。
3)培養集団を S basal 溶液中に集め、3等分する。
4)このうち2つ分の虫集団より S basal 溶液を除き、20ul の proteinase K 溶液で溶解する。
5)溶解液の 20 分の 1 量を用いて PCR を行ない、アガロースゲル電気泳動により分析する。
6)両虫集団から同じ長さの PCR 産物が得られた場合、残りの虫集団を約 25 匹ずつに分け、それぞれを培養する。
7)餌がなくなった時点で、各培養の一部の虫を proteinase K 溶液で溶解し、PCR で調べる。
8)PCR 産物が得られた場合、残りの虫より 1 匹から出発する培養を行ない、同様に調べる。
欠失変異体を得るためのスクリーニングに必要な個体数も、Tc1 挿入変異体を得る際と同様 30,000 匹程度が基準となる。上記の方法では 50 プレート分である。
遺伝子プライマーは、互いに 3kb 程度離れた位置(Tc1 の長さは含まず)に設定すると良いようである。この場合、1 - 2 kb の欠失がよく見い出される。また欠失は、Tc1 挿入部位より一方側のみで起こることが多い。
§変異のホモ接合とヘテロ接合の見分け方
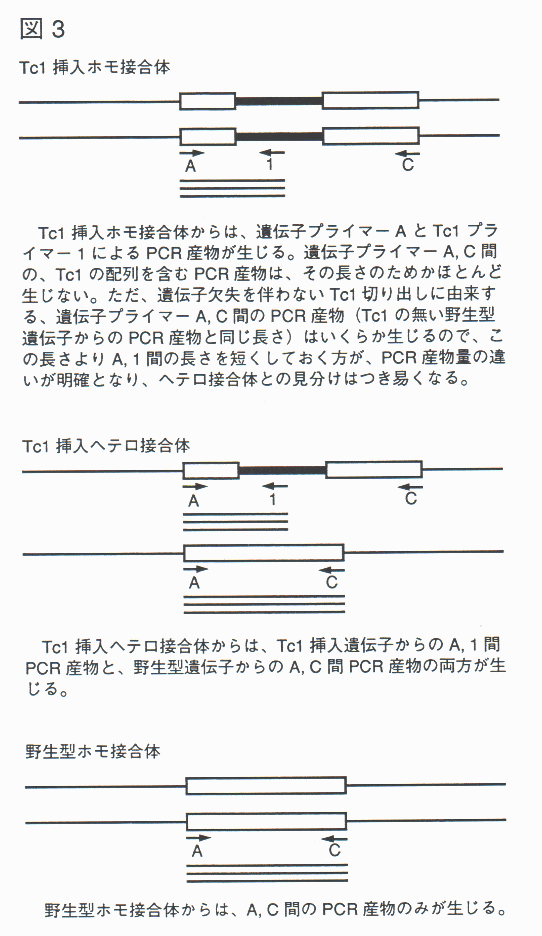
得られた変異について、ある個体がホモ接合であるかヘテロ接合であるかは、PCR により見分けることができる。ここでは、Tc1 挿入変異について説明する。欠失変異についても、同様の方法で見分けることができる。
PCR は、反応液に 3 種のプライマーを加えて行なう(図3)。この内、2 種のプライマーは Tc1 を挟む位置にある遺伝子プライマーであり、もう 1 種は Tc1 プライマーである。Tc1 挿入変異の有るゲノム DNA からは Tc1 プライマーと遺伝子プライマーの 1 つによる PCR 産物が増幅され、Tc1 挿入変異の無いゲノム DNA からは 2 種の遺伝子プライマーによる PCR 産物が増幅される。ホモ接合体からは前者の PCR 産物のみが得られ、ヘテロ接合体からは両方の PCR 産物が得られる。
以下に、具体的方法を記す。
1)プレート(餌が残っていてもよい)から虫を S basal 溶液中に移し、しばらく泳がせて虫を洗う。
2)2ul の proteinase K 溶液の入った PCR 用容器へ虫を移し、溶解させる。
3)この溶解液にPCR 反応液を加え PCR を行ない、アガロースゲル電気泳動で分析する。
ある集団が Tc1 挿入変異についてホモ接合であるかを知るには、その集団より 10 匹ほどの虫をこの方法で調べ、全てがホモ接合体であれば、その集団はホモ接合体の集団と考えられる。
§追加
Tc1 挿入変異体の分離について、ここでは触れなかったが、Tc1 挿入変異体バンクを作成する方法もある。これは、ミューテーター株の培養集団の内、ゲノム DNA 抽出に用いる虫体以外を凍結保存したものである。利点は、変異体バンクを作製してしまえば、培養を行なうことなく Tc1 挿入変異体のスクリーニングを行なえることである。しかし、 C. elegans 凍結保存後の生存率の問題などにより、培養等はここで述べたより手間がかかるものとなる。詳しくは、参考文献を参照されたい。
また最近、ゲノム DNA に欠失変異を引き起こす突然変異剤で処理した虫集団より、ターゲット遺伝子の欠失変異体を得る方法が報告された。欠失変異体の選び出しは、 Tc1 挿入変異体より欠失変異体を得るものと同じく、PCR を用いた sib-selection 法により行なう。この方法は欠失変異体を直接得ることができる点で、 Tc1 挿入変異体を経由する方法に比べ優れている。しかし、現在のところ用いられている変異剤 TMP (trimethylpsoralen) では欠失変異の起こる頻度が低いため、より多くの虫(10 倍程度)をスクリーニングしなければならないようである。この方法の詳細については、プロトコールなどが入っている WWW-site を参照されたい。
§参考文献
R.H.A.Plasterk: BioEssays, 14, 629-633 (1992) Reverse genetics of Caenorhabditis elegans.
A.M.Rushforth, B.Saari & P.Anderson: Mol. Cell Biol., 13, 902-910 (1993) Site-selected insertion of the transposon Tc1 into a Caenorhabditis elegans myosin light chain gene.
R.R.Zwaal, A.Broeks, J.van Meurs, J.T.M.Groenen & R.H.A.Plasterk: Proc. Natl. Acad. Sci. USA, 90, 7431-7435 (1993) Target-selected gene inactivation in Caenorhabditis elegans by using a frozen transposon insertion mutant bank.
H.F.Epstein & D.C.Shakes (eds.): Methods in Cell Biology Vol.48, (1995)
"Caenorhabditis elegans: Modern Biological Analysis of an Organism"
§WWW-site
http://snmc01.omrf.uokhsc.edu/revgen/RevGen.html